ProjektSTRESZCZENIEMETODYKA PLAN PRACY |
METODYKA
W okresie dwóch lat próbki zostaną pobrane z Głębi Gdańskiej oraz z dodatkowych czterech lokalizacji (Głębia Gotlandzka, Głębia Bornholmska oraz dwie stacje na Basenie Gotlandzkim) (Rys. 1.). Próbki będą pobierane w dwóch sezonach (wiosna i jesień). Rejsy będą odbywać się na statku badawczym Oceania (IO PAN). 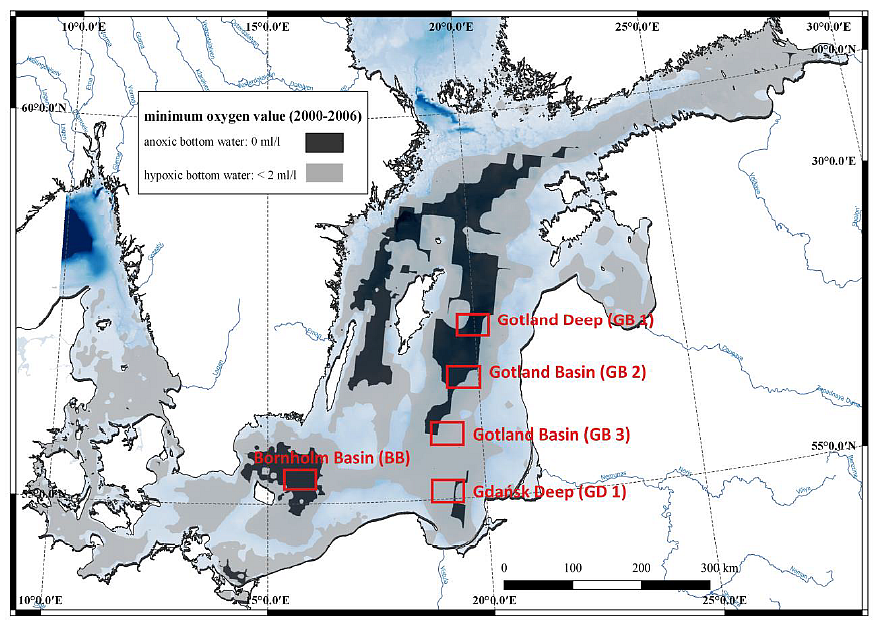
Rys. 1. Miejsca pobierania próbek i stężenie tlenu w wodach przydennych Morza Bałtyckiego w latach 2000-2006. Dane GIS dotyczące stężenia tlenu otrzymano w ramach projektu BALANCE. Próbki wody morskiej będą pobierane za pomocą rozety CTD (wyposażonej w 10 butelek Niskin), a próbki osadów za pomocą sondy typu Niemisto. Woda porowa będzie pobierana z użyciem próbników typu Rhizon. Próbki do eksperymentów inkubacyjnych, analiz metagenomicznych i metatranskryptomicznych oraz parametrów pomocniczych będą pobierane ze stref z minimalną zawartością tlenu i osadów. Strategię pobierania próbek przedstawiono na Rys. 2. Procedury eksperymentów inkubacji wody i osadu zostaną zmodyfikowane w oparciu o publikacje Dalsgaard i in. (2003, 2005, 2013), Deutsch i in. (2010) oraz Bonaglia i in. (2014, 2016). Wszystkie próbki z eksperymentów inkubacji dla wyznaczenia stosunku N2/Ar (i izotopów azotu) będą mierzone za pomocą spektrometru masowego z membraną półprzepuszczalną (ang. membrane-inlet mass spectrometry, MIMS), BAY INSTRUMENTS (Kana i wsp. 1994). Kontrola jakości będzie przeprowadzana zawsze przed wdrożeniem metody. 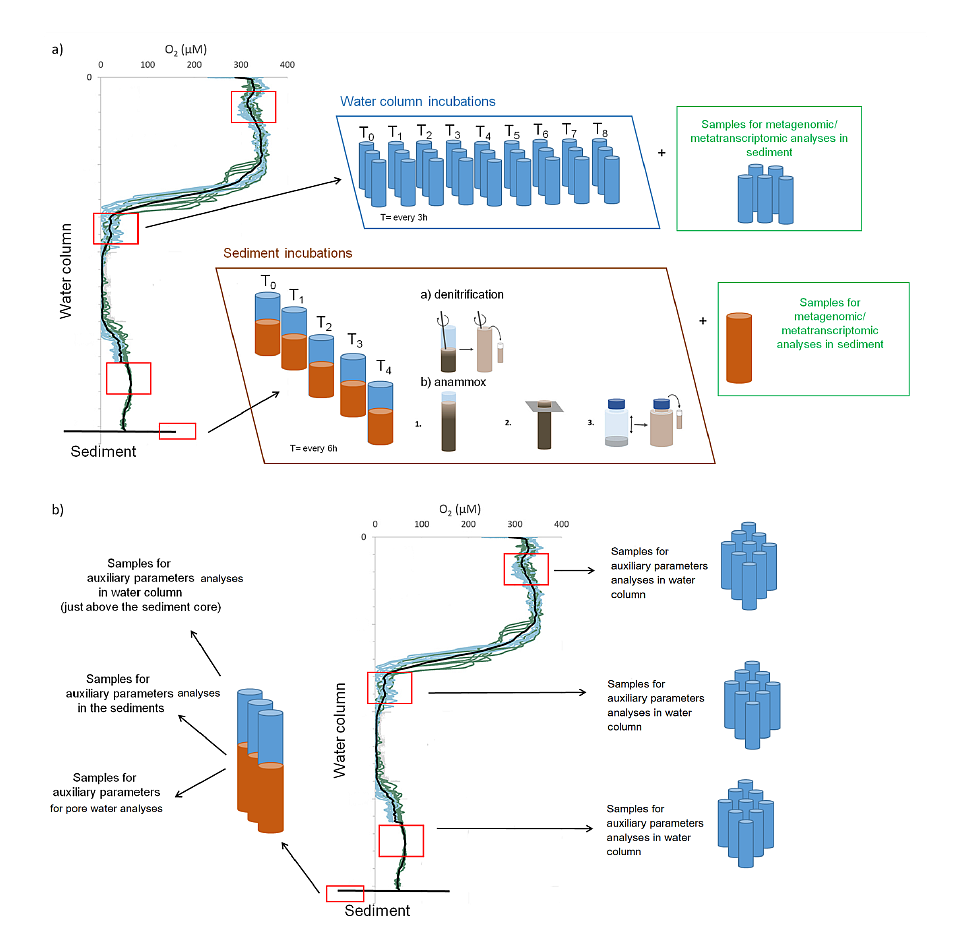
Rys. 2. Schemat strategii pobierania próbek uwzględniający a) pobieranie próbek do eksperymentów inkubacji oraz analiz metagenomicznych/metatranskryptomicznych oraz b) pobieranie próbek do analiz parametrów dodatkowych. Obecność bakteryjnego DNA w pobranych próbkach oraz brak inhibitorów zostanie potwierdzona reakcjami PCR ze starterami F27 i 1492 komplementarnymi do genu 16S rRNA. W każdej próbce przy użyciu szablonu DNA qPCR zostanie zastosowane ilościowe określenie ilości genów obiegu azotu, szczególnie tych, które mogą być aktywne w miejscach niedoboru tlenu w Morzu Bałtyckim: (np.: napa, narG, nirK, nirS, nrfA, nor, nosZ, nod, hzsA). Ponadto analiza metagenomiczna i metatranskryptomiczna posłuży do monitorowania struktury i funkcjonowania populacji mikroorganizmów. Celem tego projektu jest umieszczenie informacji o uzyskanych sekwencjach DNA w publicznych bazach danych sekwencji (np. NCBI, RDP). Całe sekwencjonowanie NGS zostanie zlecone wyspecjalizowanej firmie zewnętrznej, wybranej na podstawie aktualnej sytuacji rynkowej. Analizy parametrów pomocniczych (Tabela 1) są regularnie wykonywane w IO PAN i będą wykonywane najnowocześniejszymi metodami, a wysoka jakość uzyskanych wyników będzie oparta o certyfikaty i analizę materiałów referencyjnych. Na potrzeby proponowanego projektu nie można zastosować standardowej metody analizy biogenów ze względu zarówno na zbyt wysokie granice wykrywalności, jak i zbyt dużą objętość próbki potrzebnej do analiz. Składniki odżywcze i stosunek N2/Ar, będą mierzone równolegle i z tego powody nie będzie wystarczającej objętości próbki do każdej analizy. Dlatego w ramach tego projektu pomiary składników odżywczych będą wykonywane za pomocą zalecanego na całym świecie analizatora ciągłego przepływu (z ang. continuous flow analyzer, CFA). Zaawansowane analizy statystyczne, takie jak np. analiza głównych składowych (PCA), regresja głównych składowych (PCR), ANOVA będą wykonywane przez oprogramowanie STATISTICA. Tabela 1. Lista parametrów dodatkowych mierzonych w wyznaczonych próbkach
|
|||||||||||||||||||||||||||||||||||||||||||||||||||||||||||


